Limpieza y análisis de bases de datos
La mayor parte de los resultados de las encuestas son presentados en partes, y el vaciado de las respuestas no siempre nos permite hacer un análisis preciso de la información. Por esta razón, resulta indispensable hacer un análisis exploratorio de la base de datos para identificar respuestas incoherentes o eliminar variables o datos que no sirvan para los propósitos del investigador. En esta sección utilizamos una base de datos (ficticia) que simula la información obtenida de varios hospitales de Estados Unidos sobre sus pacientes con cáncer. El objetivo es: 1) realizar un análisis exploratorio de la información y 2) limpiar la base de datos tal que sea posible hacer análisis estadísticos más profundos.
Durante toda la sección estaremos utilizando los paquetes tidyverse y Hmisc de R, los cuales contienen comandos que simplifican la exploración y limpieza de la base de datos.
# Instalar paquetes si es necesario
# install.packages("tidyverse")
# install.packages("Hmisc")
# Leer paquetes
library(tidyverse)## ── Attaching packages ──────────────────────────── tidyverse 1.3.0 ──## ✓ ggplot2 3.3.2 ✓ purrr 0.3.4
## ✓ tibble 3.0.1 ✓ dplyr 1.0.0
## ✓ tidyr 1.1.0 ✓ stringr 1.4.0
## ✓ readr 1.3.1 ✓ forcats 0.5.0## ── Conflicts ─────────────────────────────── tidyverse_conflicts() ──
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()library(Hmisc)## Loading required package: lattice## Loading required package: survival## Loading required package: Formula##
## Attaching package: 'Hmisc'## The following objects are masked from 'package:dplyr':
##
## src, summarize## The following objects are masked from 'package:base':
##
## format.pval, unitsLimpieza de la base de datos y valores ausentes
Comenzamos leyendo la base de datos. Esta consta de 24 variables y 120 observaciones que simulan información sobre pacientes con cáncer. El archivo está en formato de valores separados por comas, .csv. Puedes descargar el archivo al disco duro de tu computadora o bien puedes leer el archivo desde su ubicación en línea. El comando de lectura puede ser el preinstalado en R: read.cvs, o bien el comando contenido en el paquete tidyverse: read_csv. Aquí utilizaremos la segunda opción, pero asegúrate de comprender las diferencias entre ambos comandos utilizando el símbolo de ayuda (por ejemplo: ?read_csv).
db <- read_csv("https://raw.githubusercontent.com/amosino/courses--econometria/master/econometria_salud/econometria_salud--datos/CancerData_P1.csv")## Parsed with column specification:
## cols(
## .default = col_double(),
## hospital = col_character(),
## docid = col_character(),
## dis_date = col_character(),
## sex = col_character(),
## familyhx = col_character(),
## smokinghx = col_character(),
## cancerstage = col_character(),
## wbc = col_character()
## )## See spec(...) for full column specifications.db## # A tibble: 120 x 24
## hospital hospid docid dis_date sex age test1 test2 pain tumorsize
## <chr> <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 UCLA 1 1-1 6-Sep-09 male 65.0 3.70 8.09 4 68.0
## 2 UCLA 1 1-1 7-Jan-11 fema… 53.9 2.63 0.803 2 64.7
## 3 UCLA 1 1-1 4-Sep-10 male 41.4 -99 2.13 3 86.4
## 4 UCLA 1 1-1 25-Jun-… male 46.8 3.89 1.35 3 53.4
## 5 UCLA 1 1-1 1-Jul-09 male 51.9 1.42 2.19 4 51.7
## 6 UCLA 1 1-1 6-Mar-09 fema… 53.8 2.29 8.61 3 78.9
## 7 UCLA 1 1-1 15-Apr-… male 54.4 8.03 7.23 4 62.9
## 8 UCLA 1 1-11 12-Jul-… fema… 47.1 0.810 2.60 5 81.2
## 9 UCLA 1 1-11 25-Jul-… fema… 59.3 -99 5.18 4 73.2
## 10 UCLA 1 1-11 12-Jul-… fema… 47.1 0.810 2.60 5 81.2
## # … with 110 more rows, and 14 more variables: co2 <dbl>, wound <dbl>,
## # mobility <dbl>, ntumors <dbl>, remission <dbl>, lungcapacity <dbl>,
## # married <dbl>, familyhx <chr>, smokinghx <chr>, cancerstage <chr>,
## # lengthofstay <dbl>, wbc <chr>, rbc <dbl>, bmi <dbl>Como primer paso, conviene hacer un análisis descriptivo de la base de datos. Esto nos permitirá no solo conocer la distribución de la muestra, si no que también nos permite detectar valores sospechosos. Para obtener los estadísticos básicos de la muestra, tenemos dos opciones. Una es utilizar la función summary(), la cual viene preinstalada en R. La otra opción es el comando describe(), el cual es parte del paquete Hmisc. Aquí preferimos la segunda opción, ya que esta nos da tablas de frecuencias para las variables discretas (que tengan menos de 20 valores), más detalle sobre los cuantiles para las variables continuas, conteos y proporciones para las variables binarias, y conteo de datos faltantes (NA).
describe(db)## db
##
## 24 Variables 120 Observations
## --------------------------------------------------------------------------------
## hospital
## n missing distinct
## 120 0 2
##
## Value UCLA UCSF
## Frequency 62 58
## Proportion 0.517 0.483
## --------------------------------------------------------------------------------
## hospid
## n missing distinct Info Mean Gmd
## 120 0 2 0.749 1.483 0.5036
##
## Value 1 2
## Frequency 62 58
## Proportion 0.517 0.483
## --------------------------------------------------------------------------------
## docid
## n missing distinct
## 120 0 22
##
## lowest : 1-1 1-100 1-11 1-21 1-22 , highest: 2-177 2-178 2-188 2-201 2-216
## --------------------------------------------------------------------------------
## dis_date
## n missing distinct
## 120 0 104
##
## lowest : 1-Jul-09 10-Jun-09 10-Jun-10 11-Apr-10 11-Dec-09
## highest: 9-Apr-10 9-Feb-09 9-Feb-10 9-Jun-10 9-May-10
## --------------------------------------------------------------------------------
## sex
## n missing distinct
## 120 0 3
##
## Value 12.2 female male
## Frequency 1 74 45
## Proportion 0.008 0.617 0.375
## --------------------------------------------------------------------------------
## age
## n missing distinct Info Mean Gmd .05 .10
## 120 0 117 1 53.59 11.92 40.86 41.52
## .25 .50 .75 .90 .95
## 47.75 51.83 55.01 58.61 59.65
##
## lowest : 34.19229 35.31930 37.25225 39.61641 40.03724
## highest: 63.93238 64.16432 64.96824 65.80417 357.89001
##
## Value 35 40 45 50 55 60 65 360
## Frequency 3 10 16 38 35 13 4 1
## Proportion 0.025 0.083 0.133 0.317 0.292 0.108 0.033 0.008
##
## For the frequency table, variable is rounded to the nearest 5
## --------------------------------------------------------------------------------
## test1
## n missing distinct Info Mean Gmd .05 .10
## 120 0 111 1 -1.989 14.11 -99.0000 0.5809
## .25 .50 .75 .90 .95
## 1.5597 3.1065 5.7067 7.9931 9.5583
##
## lowest : -99.0000000 0.1048958 0.1927608 0.4293420 0.5155185
## highest: 9.7981329 10.2903990 10.4685400 11.0714440 12.4163920
##
## Value -99 0 1 2 3 4 5 6 7 8 9
## Frequency 7 3 20 17 21 12 8 11 6 7 1
## Proportion 0.058 0.025 0.167 0.142 0.175 0.100 0.067 0.092 0.050 0.058 0.008
##
## Value 10 11 12
## Frequency 5 1 1
## Proportion 0.042 0.008 0.008
##
## For the frequency table, variable is rounded to the nearest 1
## --------------------------------------------------------------------------------
## test2
## n missing distinct Info Mean Gmd .05 .10
## 120 0 111 1 -1.226 14.59 -99.0000 0.7881
## .25 .50 .75 .90 .95
## 2.2494 4.1620 6.1657 8.9973 10.9861
##
## lowest : -99.0000000 0.5807591 0.6179262 0.6571499 0.7485080
## highest: 11.4315750 12.4493730 14.2252270 14.5365420 17.2275810
##
## Value -99 1 2 3 4 5 6 7 8 9 10
## Frequency 7 15 12 19 16 15 9 6 6 6 1
## Proportion 0.058 0.125 0.100 0.158 0.133 0.125 0.075 0.050 0.050 0.050 0.008
##
## Value 11 12 14 15 17
## Frequency 4 1 1 1 1
## Proportion 0.033 0.008 0.008 0.008 0.008
##
## For the frequency table, variable is rounded to the nearest 1
## --------------------------------------------------------------------------------
## pain
## n missing distinct Info Mean Gmd
## 120 0 9 0.961 5.325 1.742
##
## lowest : 1 2 3 4 5, highest: 5 6 7 8 9
##
## Value 1 2 3 4 5 6 7 8 9
## Frequency 1 2 11 21 33 25 18 5 4
## Proportion 0.008 0.017 0.092 0.175 0.275 0.208 0.150 0.042 0.033
## --------------------------------------------------------------------------------
## tumorsize
## n missing distinct Info Mean Gmd .05 .10
## 120 0 117 1 70.08 12.92 53.48 55.97
## .25 .50 .75 .90 .95
## 62.18 68.23 77.48 84.06 92.54
##
## lowest : 49.09861 50.28009 50.50217 51.65727 53.19937
## highest: 98.05510 98.32850 98.70570 102.69671 109.00956
## --------------------------------------------------------------------------------
## co2
## n missing distinct Info Mean Gmd .05 .10
## 120 0 115 1 -0.8901 5.03 1.390 1.440
## .25 .50 .75 .90 .95
## 1.495 1.586 1.686 1.756 1.793
##
## lowest : -98.000000 1.327440 1.362927 1.372113 1.391310
## highest: 1.810847 1.820266 1.905606 1.920025 1.942401
##
## Value -98 1 2
## Frequency 3 29 88
## Proportion 0.025 0.242 0.733
##
## For the frequency table, variable is rounded to the nearest 1
## --------------------------------------------------------------------------------
## wound
## n missing distinct Info Mean Gmd
## 120 0 8 0.958 5.592 1.767
##
## lowest : 2 3 4 5 6, highest: 5 6 7 8 9
##
## Value 2 3 4 5 6 7 8 9
## Frequency 5 10 12 23 36 22 10 2
## Proportion 0.042 0.083 0.100 0.192 0.300 0.183 0.083 0.017
## --------------------------------------------------------------------------------
## mobility
## n missing distinct Info Mean Gmd
## 120 0 8 0.969 6.033 2.264
##
## lowest : 2 3 4 5 6, highest: 5 6 7 8 9
##
## Value 2 3 4 5 6 7 8 9
## Frequency 6 4 19 19 28 15 5 24
## Proportion 0.050 0.033 0.158 0.158 0.233 0.125 0.042 0.200
## --------------------------------------------------------------------------------
## ntumors
## n missing distinct Info Mean Gmd .05 .10
## 120 0 10 0.981 3.15 3.101 0 0
## .25 .50 .75 .90 .95
## 1 2 5 8 9
##
## lowest : 0 1 2 3 4, highest: 5 6 7 8 9
##
## Value 0 1 2 3 4 5 6 7 8 9
## Frequency 23 21 19 9 12 11 7 5 5 8
## Proportion 0.192 0.175 0.158 0.075 0.100 0.092 0.058 0.042 0.042 0.067
## --------------------------------------------------------------------------------
## remission
## n missing distinct Info Sum Mean Gmd
## 120 0 2 0.683 42 0.35 0.4588
##
## --------------------------------------------------------------------------------
## lungcapacity
## n missing distinct Info Mean Gmd .05 .10
## 120 0 96 0.995 -18.33 31.26 -99.0000 -99.0000
## .25 .50 .75 .90 .95
## 0.5142 0.7457 0.8721 0.9573 0.9790
##
## lowest : -99.0000000 -98.0000000 0.2949074 0.3264440 0.3450253
## highest: 0.9856416 0.9864109 0.9924814 0.9940955 0.9982018
##
## Value -99.0 -98.0 0.2 0.4 0.6 0.8 1.0
## Frequency 20 3 1 6 21 46 23
## Proportion 0.167 0.025 0.008 0.050 0.175 0.383 0.192
##
## For the frequency table, variable is rounded to the nearest 0.2
## --------------------------------------------------------------------------------
## married
## n missing distinct Info Sum Mean Gmd
## 120 0 2 0.697 76 0.6333 0.4683
##
## --------------------------------------------------------------------------------
## familyhx
## n missing distinct
## 120 0 3
##
## Value -99 no yes
## Frequency 6 97 17
## Proportion 0.050 0.808 0.142
## --------------------------------------------------------------------------------
## smokinghx
## n missing distinct
## 120 0 4
##
## Value -99 current former never
## Frequency 6 26 22 66
## Proportion 0.050 0.217 0.183 0.550
## --------------------------------------------------------------------------------
## cancerstage
## n missing distinct
## 120 0 4
##
## Value I II III IV
## Frequency 40 54 17 9
## Proportion 0.333 0.450 0.142 0.075
## --------------------------------------------------------------------------------
## lengthofstay
## n missing distinct Info Mean Gmd
## 120 0 6 0.933 5.308 1.271
##
## lowest : 3 4 5 6 7, highest: 4 5 6 7 8
##
## Value 3 4 5 6 7 8
## Frequency 5 29 29 40 15 2
## Proportion 0.042 0.242 0.242 0.333 0.125 0.017
## --------------------------------------------------------------------------------
## wbc
## n missing distinct
## 120 0 116
##
## lowest : 3671.880371 4176.054199 4201.741211 4238.355957 4331.902344
## highest: 7999.091309 8340.71582 8415.605469 8567.246094 not assessed
## --------------------------------------------------------------------------------
## rbc
## n missing distinct Info Mean Gmd .05 .10
## 120 0 117 1 4.97 0.2885 4.526 4.606
## .25 .50 .75 .90 .95
## 4.825 4.978 5.150 5.285 5.353
##
## lowest : 4.359662 4.436482 4.456108 4.465468 4.470100
## highest: 5.441604 5.442614 5.459067 5.502604 5.535052
## --------------------------------------------------------------------------------
## bmi
## n missing distinct Info Mean Gmd .05 .10
## 120 0 117 1 29.38 7.278 20.71 22.07
## .25 .50 .75 .90 .95
## 24.51 27.82 34.31 37.75 40.71
##
## lowest : 18.44992 18.68505 20.07485 20.17994 20.49195
## highest: 42.84858 44.04223 46.50746 52.30723 58.00000
## --------------------------------------------------------------------------------A simple vista podemos detectar algunos valores sospechosos. Por ejemplo:
describe(db[,c("age", "sex", "test1")])## db[, c("age", "sex", "test1")]
##
## 3 Variables 120 Observations
## --------------------------------------------------------------------------------
## age
## n missing distinct Info Mean Gmd .05 .10
## 120 0 117 1 53.59 11.92 40.86 41.52
## .25 .50 .75 .90 .95
## 47.75 51.83 55.01 58.61 59.65
##
## lowest : 34.19229 35.31930 37.25225 39.61641 40.03724
## highest: 63.93238 64.16432 64.96824 65.80417 357.89001
##
## Value 35 40 45 50 55 60 65 360
## Frequency 3 10 16 38 35 13 4 1
## Proportion 0.025 0.083 0.133 0.317 0.292 0.108 0.033 0.008
##
## For the frequency table, variable is rounded to the nearest 5
## --------------------------------------------------------------------------------
## sex
## n missing distinct
## 120 0 3
##
## Value 12.2 female male
## Frequency 1 74 45
## Proportion 0.008 0.617 0.375
## --------------------------------------------------------------------------------
## test1
## n missing distinct Info Mean Gmd .05 .10
## 120 0 111 1 -1.989 14.11 -99.0000 0.5809
## .25 .50 .75 .90 .95
## 1.5597 3.1065 5.7067 7.9931 9.5583
##
## lowest : -99.0000000 0.1048958 0.1927608 0.4293420 0.5155185
## highest: 9.7981329 10.2903990 10.4685400 11.0714440 12.4163920
##
## Value -99 0 1 2 3 4 5 6 7 8 9
## Frequency 7 3 20 17 21 12 8 11 6 7 1
## Proportion 0.058 0.025 0.167 0.142 0.175 0.100 0.067 0.092 0.050 0.058 0.008
##
## Value 10 11 12
## Frequency 5 1 1
## Proportion 0.042 0.008 0.008
##
## For the frequency table, variable is rounded to the nearest 1
## --------------------------------------------------------------------------------Otra de las ventajas que tiene el comando describe(), es que este se puede graficar con el comando plot(). Este comando aplicado a describe() resulta en gráficas de puntos para las variables categóricas (o discretas) e histogramas para las variables continuas. Además, las gráficas incluyen un indicador de color para mostrar el número de NA que contiene cada variable.
plot(describe(db))## $Categorical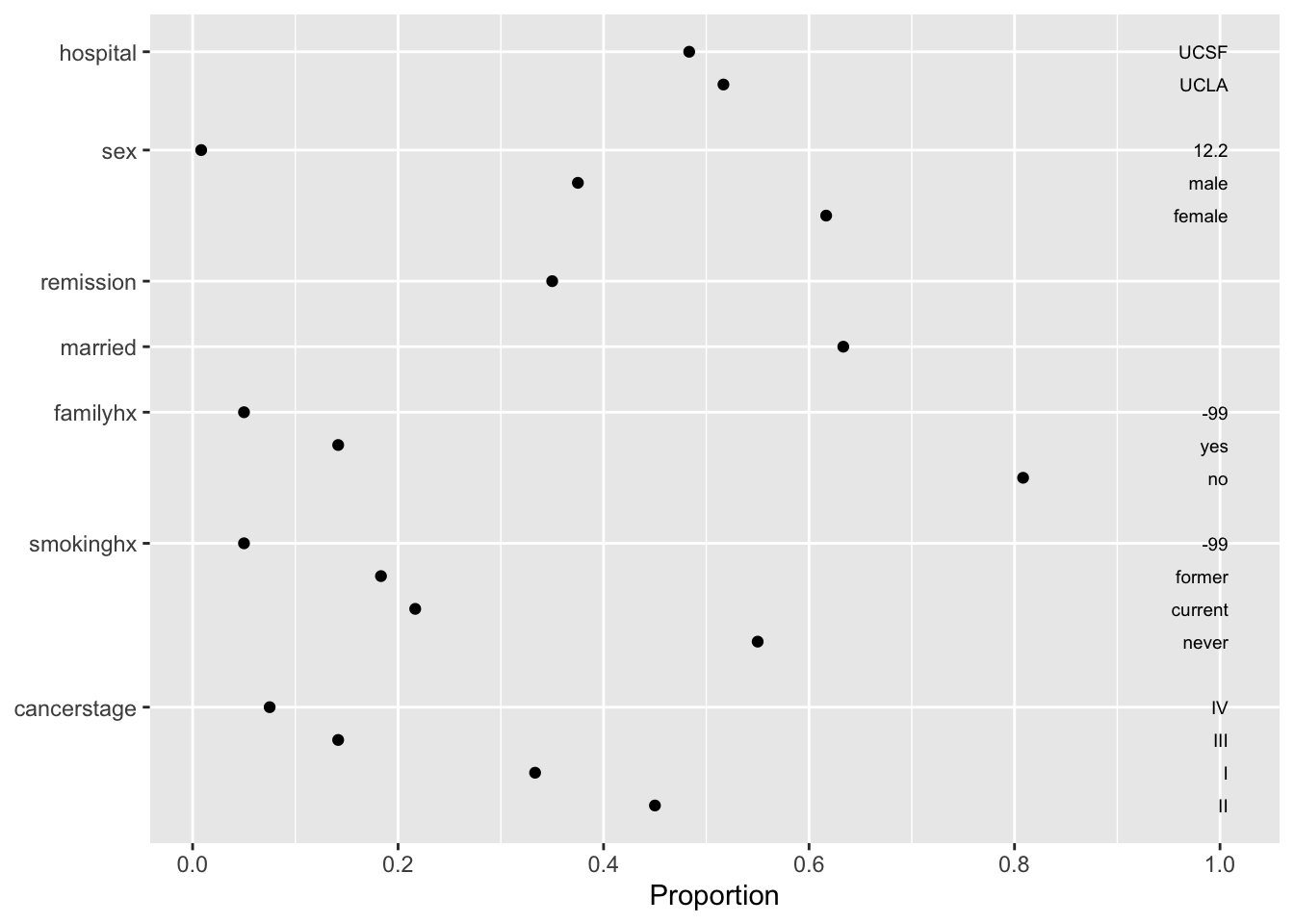
##
## $Continuous
Normalmente, las gráficas tienden a revelar más información sobre los valores sospechosos que las tablas de datos. Sin embargo, ambas revelan la misma información. En nuestro caso:
- La variable
edadtiene un valor máximo de 357.89001. - La varible
sexmuestra una categoría “12.2”. - Algunas variables, como
test1,test2,lungcapacity, entre otras tienen valores de -99 (el cual, usualmente, corresponde a “no sabe” o “ausente”). - Algunas variables, como
co2, ylungcapacitytienen valores de -98 (el cual, usualmente, corresponde a “no quiso contestar”). wbcparece ser una variable numérica, pero, debido a que tiene valores “not assesed”, R la interpreta como si se tratase de una cadena de caracteres.
Para que la base de datos sea utilizable, podemos reemplazar todos estos valores sospechosos por NA. Por supuesto que también es posible reemplazar los valores sopechosos con sus valores verdaderos, pero esto normalmente no es posible porque esta es información a la que normalmente no tenemos acceso. La siguiente secuencia de comandos haría el trabajo:
db$age[db$age<18|db$age>120] <- NA
db$sex[db$sex == "12.2"] <- NA
db[db==-99] <- NA
db[db==-98] <- NA
db[db=="not assesed"] <- NAVolvemos a utilizar los comandos describe() y plot() para ver cómo ha cambiado la gráfica descriptiva. Mucho mejor, ¿no?
plot(describe(db))## $Categorical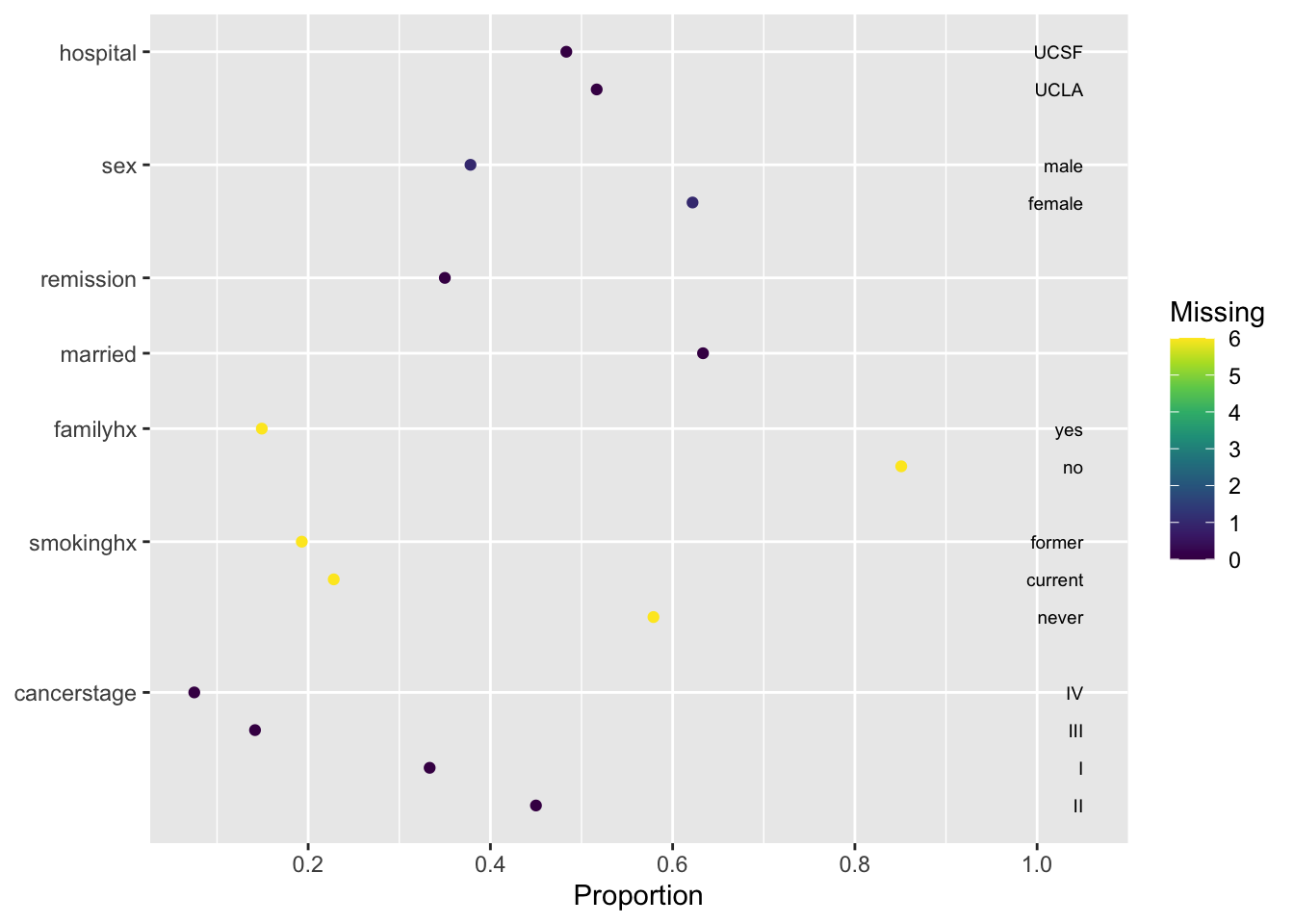
##
## $Continuous
Otros comandos útiles
Otros comandos que pueden ayudarnos a limpiar la base de datos son: filter(), select(), arrange(), mutate() y summarize(), los cuales son parte del paquete tidyverse. El uso de estos comandos tiene algunas ventajas. Especificamente, estas funciones simplifican el código, ya que solo hay que nombrar la base de datos una vez y proporcionar las variables como argumentos. Además, las variables no tienen que escribirse entre comillas.
filter()
El comando filter() nos permite elegir una submuestra usando operadores lógicos. Estos operadores pueden ser:
==: Igualdad.!=: Desigualdad.>,>=: Mayor que o mayor o igual que.<,<=: Menor que o menor o igual que.&: Y.|: O.- %in%: En.
is.na: es igual aNA.near(): Igualdad para un valor con decimales. Puede modificarse la tolerancia.
Un ejemplo de uso para el comando filter() es elegir todas las personas de género femenino con un grado de dolor mayor o igual a 9:
dfhp <- filter(db, sex=="female", pain>=9)¿Cómo encontrarías este subgrupo de pacientes sin utilizar filter()?
select()
El comando select() se utiliza para mantener solo las variables que nos interesan. Por ejemplo, si deseamos mantener solo las variables hospid, docid, age y cancerstage:
db_small <- select(db, hospid, docid, age, cancerstage)
db_small## # A tibble: 120 x 4
## hospid docid age cancerstage
## <dbl> <chr> <dbl> <chr>
## 1 1 1-1 65.0 II
## 2 1 1-1 53.9 II
## 3 1 1-1 41.4 I
## 4 1 1-1 46.8 II
## 5 1 1-1 51.9 I
## 6 1 1-1 53.8 II
## 7 1 1-1 54.4 II
## 8 1 1-11 47.1 I
## 9 1 1-11 59.3 IV
## 10 1 1-11 47.1 I
## # … with 110 more rowsTambién podemos seleccionar un grupo de variables con nombres similares. Esto se hace con las funciones:
starts_with(x): Comienza con.ends_with(x): Termina con.contains(x): Contiene.matches(re): Encuentra la expresión regularesre.num_range(prefijo, rango): Encuentra todos los nombres de variables que contenganprefijoy un elemento derango.
Por ejemplo: seleccionar todas las variables que comiencen con hosp y obtener las variables test1 y test2 usando num_range().
# Obtener las variables que comiencen con hosp
dbhosp <- select(db, starts_with("hosp"))
names(dbhosp)## [1] "hospital" "hospid"# Obtener las variables test1 y tes2
dbhosp <- select(db, num_range("test", 1:2))
names(dbhosp)## [1] "test1" "test2"arrange()
El comando arrange() nos permite ordenar las filas de una base de datos de acuerdo a los criterios especificados por el usuario. El comando arrange() ordena automáticamente en orden ascendente, pero se puede ordenar el orden descendente si utilizamos el argumento desc. Por ejemplo, podemos ordenar nuestra base de datos primero hombres, luego mujeres y luego por edades, primero lo más jóvenes:
arrange(db, desc(sex), age)## # A tibble: 120 x 24
## hospital hospid docid dis_date sex age test1 test2 pain tumorsize co2
## <chr> <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 UCSF 2 2-178 19-Apr-… male 34.2 4.63 3.26 3 65.5 1.64
## 2 UCLA 1 1-58 9-Feb-10 male 35.3 2.14 12.4 6 60.5 1.56
## 3 UCSF 2 2-121 20-Apr-… male 40.1 NA NA 5 73.5 1.65
## 4 UCLA 1 1-1 4-Sep-10 male 41.4 NA 2.13 3 86.4 1.45
## 5 UCSF 2 2-121 22-Aug-… male 41.5 2.88 6.80 5 60.2 1.50
## 6 UCSF 2 2-163 19-Apr-… male 42.5 1.63 0.749 4 54.0 1.72
## 7 UCLA 1 1-11 19-Aug-… male 43.7 10.5 5.89 7 61.3 1.49
## 8 UCLA 1 1-1 25-Jun-… male 46.8 3.89 1.35 3 53.4 1.57
## 9 UCLA 1 1-21 22-Apr-… male 48.0 3.39 NA 8 79.6 1.55
## 10 UCSF 2 2-178 12-Mar-… male 48.9 4.15 3.04 7 71.8 1.47
## # … with 110 more rows, and 13 more variables: wound <dbl>, mobility <dbl>,
## # ntumors <dbl>, remission <dbl>, lungcapacity <dbl>, married <dbl>,
## # familyhx <chr>, smokinghx <chr>, cancerstage <chr>, lengthofstay <dbl>,
## # wbc <chr>, rbc <dbl>, bmi <dbl>mutate()
El comando mutate() nos permite transformar muchas variables en un solo paso sin tener que espcificar una y otra vez el nombre de la base de datos a la que pertenecen. Algunas de las transformaciones que pueden hacerse en R son:
log(): Logaritmo.cut(): Corta una variable continua en intervalos.scale(): Estandariza una variable (resta la media y divide entre su desviación estándar.lag()ylead(): Rezaga o adelanta una variable.cumsum(): Suma acumulada.rowMeans()yrowSums(): Promedios y sumas de varias columnas.recode(): Recodifica valores.
Como ejemplo, supongamos que deseamos crear una variable de edad, agecat, por intervalos de 10 años y una variable binaria, highpain, que indique las personas que tienen más dolor que el promedio:
db <- mutate(db, agecat=cut(age, breaks=c(30,40,50,60,70,120)), highpain=pain>mean(pain))
table(db$agecat, db$highpain)##
## FALSE TRUE
## (30,40] 2 2
## (40,50] 27 15
## (50,60] 37 32
## (60,70] 2 2
## (70,120] 0 0Unir y combinar bases de datos
En ocasiones las bases de datos están divididas en varias partes. Cada parte puede contener las mismas variables para nuevos individuos, o cada parte puede contener nuevas variables para los mismos individuos. En el primer caso, lo que tenemos que hacer es unir las diferentes partes de las bases de datos, y en el segundo lo que tenemos que hacer es combinarlas.
Unir bases de datos
Considera una segunda parte de la base de datos que hemos considerado hasta ahora. Tómate el tiempo para analizarla. Nota que esta parte contiene prácticamente las mismas variables que la base de datos original, pero contiene nuevos individuos. Las diferencias entre las dos bases de datos resultan de las manipulaciones que hemos hecho arriba a la base de datos original.
db2 <- read_csv("https://raw.githubusercontent.com/amosino/courses--econometria/master/econometria_salud/econometria_salud--datos/CancerData_P2.csv")## Parsed with column specification:
## cols(
## .default = col_double(),
## hospital = col_character(),
## docid = col_character(),
## dis_date = col_character(),
## sex = col_character(),
## familyhx = col_character(),
## smokinghx = col_character(),
## cancerstage = col_character(),
## wbc = col_character()
## )## See spec(...) for full column specifications.db2## # A tibble: 111 x 24
## hospital hospid docid dis_date sex age test1 test2 pain tumorsize
## <chr> <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Cedars-… 3 3-227 1-Oct-09 male 54.2 3.87 1.37 6 69.8
## 2 Cedars-… 3 3-227 18-Feb-… fema… 55.1 7.76 4.50 8 68.0
## 3 Cedars-… 3 3-227 30-Jun-… fema… 58.1 5.73 -99 7 65.1
## 4 Cedars-… 3 3-227 15-Nov-… fema… 51.5 8.58 4.61 9 71.4
## 5 Cedars-… 3 3-227 17-Feb-… fema… 54.6 6.70 2.64 4 69.5
## 6 Cedars-… 3 3-227 22-Dec-… male 49.1 2.87 5.46 5 89.7
## 7 Cedars-… 3 3-227 15-Apr-… fema… 59.8 9.60 3.37 6 73.1
## 8 Cedars-… 3 3-241 17-Apr-… fema… 49.7 0.175 8.69 3 80.6
## 9 Cedars-… 3 3-241 12-Oct-… male 65.6 6.08 6.79 5 55.1
## 10 Cedars-… 3 3-241 30-Oct-… male 57.4 6.21 14.0 8 61.3
## # … with 101 more rows, and 14 more variables: co2 <dbl>, wound <dbl>,
## # mobility <dbl>, ntumors <dbl>, remission <dbl>, lungcapacity <dbl>,
## # married <dbl>, familyhx <chr>, smokinghx <chr>, cancerstage <chr>,
## # lengthofstay <dbl>, wbc <chr>, rbc <dbl>, bmi <dbl>El objetivo es unir las bases de datos. Para esto podemos utilizar el comando preinstalado rbind(), o bien el comando bind_rowns() que es parte del paquete tidyverse. Existen algunas diferencias entre ambos métodos. Por ejemplo, si en una de las bases de datos que vamos a unir existen columnas que en la otra no rbind() resultará en un error, mientras que bind_rows() creará las nuevas columnas y rellenará los datos faltantes con NA. Otra diferencia es que si la misma columna en ambas bases de datos contiene datos de diferentes tipos (por ejemplo, en una base de datos son caracteres y en la otra son datos numéricos), rbind() convertirá los datos de la base de datos unificada en datos del mismo tipo siguiendo el orden: datos lógicos, datos numéricos enteros, datos numéricos con decimales, y, finalmente, caracteres; en cambio bind_rows() resultará en un error. Aquí utilizamos el comando bind_rows(), pero explora cómo hacer la unión de las bases usando rbind().
db_union <- bind_rows(db, db2, .id="source")
db_union## # A tibble: 231 x 27
## source hospital hospid docid dis_date sex age test1 test2 pain
## <chr> <chr> <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 1 UCLA 1 1-1 6-Sep-09 male 65.0 3.70 8.09 4
## 2 1 UCLA 1 1-1 7-Jan-11 fema… 53.9 2.63 0.803 2
## 3 1 UCLA 1 1-1 4-Sep-10 male 41.4 NA 2.13 3
## 4 1 UCLA 1 1-1 25-Jun-… male 46.8 3.89 1.35 3
## 5 1 UCLA 1 1-1 1-Jul-09 male 51.9 1.42 2.19 4
## 6 1 UCLA 1 1-1 6-Mar-09 fema… 53.8 2.29 8.61 3
## 7 1 UCLA 1 1-1 15-Apr-… male 54.4 8.03 7.23 4
## 8 1 UCLA 1 1-11 12-Jul-… fema… 47.1 0.810 2.60 5
## 9 1 UCLA 1 1-11 25-Jul-… fema… 59.3 NA 5.18 4
## 10 1 UCLA 1 1-11 12-Jul-… fema… 47.1 0.810 2.60 5
## # … with 221 more rows, and 17 more variables: tumorsize <dbl>, co2 <dbl>,
## # wound <dbl>, mobility <dbl>, ntumors <dbl>, remission <dbl>,
## # lungcapacity <dbl>, married <dbl>, familyhx <chr>, smokinghx <chr>,
## # cancerstage <chr>, lengthofstay <dbl>, wbc <chr>, rbc <dbl>, bmi <dbl>,
## # agecat <fct>, highpain <lgl>Nota que en la unión hemos utilizado el argumento .id="source". Este creará una nueva columna al inicio de la base de datos que nos indica el origen de cada fila. En este caso, un valor de 1 indica que esa observación proviene de la primer base de datos (db) y un valor de 2 de la segunda base de datos (db2).
Combinar bases de datos
Ahora supongamos que una tercera base de datos contiene nuevas variables, pero (virtualmente) los mismos individuos. En este caso, las bases de datos pueden combinarse si existe alguna o algunas variales que identifiquen a los individuos.
db3 <- read_csv("https://raw.githubusercontent.com/amosino/courses--econometria/master/econometria_salud/econometria_salud--datos/CancerData_D1.csv")## Parsed with column specification:
## cols(
## docid = col_character(),
## experience = col_double(),
## school = col_character(),
## lawsuits = col_double(),
## medicaid = col_double()
## )db3## # A tibble: 40 x 5
## docid experience school lawsuits medicaid
## <chr> <dbl> <chr> <dbl> <dbl>
## 1 1-1 25 average 3 0.606
## 2 1-11 10 top 0 0.605
## 3 1-21 21 average 3 0.483
## 4 1-22 22 top 3 0.483
## 5 1-33 16 top 0 0.584
## 6 1-48 23 average 3 0.219
## 7 1-57 21 average 1 0.405
## 8 1-58 21 average 1 0.405
## 9 1-72 24 average 4 0.522
## 10 1-73 14 average 1 0.522
## # … with 30 more rowsAl igual que en la sección anterior, existen al menos dos métodos para combinar las bases de datos en R. Una de ellas es utilizar el comando preinstalado merge() o bien el comando dplyr() del paquete tidyverse. En realidad no existen diferencias importantes entre ambas opciones, aunque merge() tiende a ser un poco más lento computacionalmente que dplyr(). Este último tiene tres diferentes versiones:
inner_join(x,y): regresa todas las filas de la base de datosxque coinciden con las filas de la base de datosy.left_join(x,y): regresa todas las filas de la base de datosx. Las filas dexque no coincidan con las filas de la base de datosyse remplazan conNA. Las filas de la base de datosyque no coincidan con las de la basexno se consideran.full_join(x,y): regresa todas las filas de la base de datosxy todas las filas de la base de datosy. Las filas dexque no coincidan con las filas de la base de datosyy las filas deyque no coincidan con la base de datosxse remplazan conNA.
Tómate el tiempo para explorar las tres posibilidades. Aquí utilizamos full_join() para mostrar el uso de este comando. Nota que R elige la variable docid para combinar las bases de datos de manera automática. Sin embargo, también es posible utilizar el argumento by para elegir la variable para combinar las bases de forma “manual”.
db_full <- full_join(db_union, db3)## Joining, by = "docid"db_full## # A tibble: 233 x 31
## source hospital hospid docid dis_date sex age test1 test2 pain
## <chr> <chr> <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 1 UCLA 1 1-1 6-Sep-09 male 65.0 3.70 8.09 4
## 2 1 UCLA 1 1-1 7-Jan-11 fema… 53.9 2.63 0.803 2
## 3 1 UCLA 1 1-1 4-Sep-10 male 41.4 NA 2.13 3
## 4 1 UCLA 1 1-1 25-Jun-… male 46.8 3.89 1.35 3
## 5 1 UCLA 1 1-1 1-Jul-09 male 51.9 1.42 2.19 4
## 6 1 UCLA 1 1-1 6-Mar-09 fema… 53.8 2.29 8.61 3
## 7 1 UCLA 1 1-1 15-Apr-… male 54.4 8.03 7.23 4
## 8 1 UCLA 1 1-11 12-Jul-… fema… 47.1 0.810 2.60 5
## 9 1 UCLA 1 1-11 25-Jul-… fema… 59.3 NA 5.18 4
## 10 1 UCLA 1 1-11 12-Jul-… fema… 47.1 0.810 2.60 5
## # … with 223 more rows, and 21 more variables: tumorsize <dbl>, co2 <dbl>,
## # wound <dbl>, mobility <dbl>, ntumors <dbl>, remission <dbl>,
## # lungcapacity <dbl>, married <dbl>, familyhx <chr>, smokinghx <chr>,
## # cancerstage <chr>, lengthofstay <dbl>, wbc <chr>, rbc <dbl>, bmi <dbl>,
## # agecat <fct>, highpain <lgl>, experience <dbl>, school <chr>,
## # lawsuits <dbl>, medicaid <dbl>Otros problemas con las bases de datos
Para terminar, veremos algunos otros problemas que podríamos enfrentar cuando analizamos bases de datos. Por ejemplo, quizás sea necesario reestructurar la base de datos de tal forma que esta pueda utilizarse para realizar un análisis estadístico o econométrico. También suele ser muy frecuente encontrar múltiples variables en la misma celda, o bien el valor de alguna variable separado en varias celdas. El objetivo de esta sección es tratar estos casos.
gather()
Considera la siguiente base de datos (ficticia), que contiene el número de egresados por año en diferentes departamentos universitarios:
db_dept <- read_csv("https://raw.githubusercontent.com/amosino/courses--econometria/master/econometria_salud/econometria_salud--datos/DeptsData.csv")## Parsed with column specification:
## cols(
## id = col_character(),
## `2015` = col_double(),
## `2016` = col_double(),
## `2017` = col_double()
## )db_dept## # A tibble: 3 x 4
## id `2015` `2016` `2017`
## <chr> <dbl> <dbl> <dbl>
## 1 biology 207 211 259
## 2 math 96 75 99
## 3 physics 112 126 125Supongamos que nuestro interés es saber si el índice de egreso ha crecido a través del tiempo. Nota que el año debería ser la variable explicativa en nuestro análisis. Sin embargo, la base de datos la incluye como nombre de las variables. Para casos como este, el comando gather() nos permite restructurar la base de datos de tal forma que:
- Una de las columnas de la base reestructurada se forme a partir de los nombres actuales de las variables. El nombre de esta columna se elige con el argumento
key. - Los valores de las columnas originales se apilen en una sola columna. El nombre de esta columna se elige con el argumento
value.
El comando gather() apila todas las columnas de la base de datos de manera automática. Sin embargo, existen algunas columnas que no cambian con los valores en la nueva columna con el nombre key; estas no deberían apilarse. Este es el caso, por ejemplo, de la actual variable id, el nombre de los departamentos no cambia con el año analizado. Para especificar cuáles variables cambian con key podemos ya sea espcificar cuáles son las columnas que deben apilarse, o bien especificar cuáles columnas no deben apilarse utilizando el símbolo -.
db_deptap <- gather(db_dept, key="year", value="grad", -id)
db_deptap## # A tibble: 9 x 3
## id year grad
## <chr> <chr> <dbl>
## 1 biology 2015 207
## 2 math 2015 96
## 3 physics 2015 112
## 4 biology 2016 211
## 5 math 2016 75
## 6 physics 2016 126
## 7 biology 2017 259
## 8 math 2017 99
## 9 physics 2017 125¿Qué hubiera pasado si no usamos el argumento -id?
spread()
Ahora considera la siguiente base de datos, que contiene información sobre diferentes tipos de gusanos clasificados por edad, tamaño y peso. Estas características, sin embargo, están apiladas en la misma columna:
db_worms <- read_csv("https://raw.githubusercontent.com/amosino/courses--econometria/master/econometria_salud/econometria_salud--datos/WormsData.csv")## Parsed with column specification:
## cols(
## worm = col_double(),
## feature = col_character(),
## measure = col_double()
## )db_worms## # A tibble: 9 x 3
## worm feature measure
## <dbl> <chr> <dbl>
## 1 1 age 5
## 2 1 length 3.2
## 3 1 weight 4.1
## 4 2 age 4
## 5 2 length 2.6
## 6 2 weight 3.5
## 7 3 age 5
## 8 3 length 3.6
## 9 3 weight 5.5Supongamos que nuestro interés es analizar la relación que existe entre la edad, el peso y el tamaño del gusano. En este caso, necesitamos que cada una de estas características constituya una columna independiente. Esta es la función del comando spread(). Los argumentos del comando spread() son nuevamente keyy value. En este caso, key es la columna que será convertida en el nombre de las variables, y value es la columna de valores que será organizada en las nuevas columnas.
worms_spread <- spread(db_worms, key=feature, value=measure)
worms_spread## # A tibble: 3 x 4
## worm age length weight
## <dbl> <dbl> <dbl> <dbl>
## 1 1 5 3.2 4.1
## 2 2 4 2.6 3.5
## 3 3 5 3.6 5.5separate() y unite()
Finalmente, considera la siguiente base de datos (automáticamente cargada con el paquete tidyverse):
db_table <- table5
db_table## # A tibble: 6 x 4
## country century year rate
## * <chr> <chr> <chr> <chr>
## 1 Afghanistan 19 99 745/19987071
## 2 Afghanistan 20 00 2666/20595360
## 3 Brazil 19 99 37737/172006362
## 4 Brazil 20 00 80488/174504898
## 5 China 19 99 212258/1272915272
## 6 China 20 00 213766/1280428583La tabla de datos que hemos cargado tiene al menos dos defectos. Primero, la variable rate en realidad contiene dos valores: número de casos y población. Segundo, las columnas century y year constituyen el año. El primer problema se resuelve con el comando separate(), el segundo con el comando unite().
Como su nombre lo indica, el comando separate() divide una columna en varias. Sus argumentos son: 1) col, el nombre de la columna a dividir, 2) into, el nombre de las nuevas columnas, 3) sep, el caracter que indica dónde separar una columna y 4) remove, que indica si las columnas concatenadas serán eliminada o no de la base de datos (pre establecido en TRUE.)
table_final <- separate(db_table, col=rate, into=c("cases", "population"), sep="/")
table_final## # A tibble: 6 x 5
## country century year cases population
## <chr> <chr> <chr> <chr> <chr>
## 1 Afghanistan 19 99 745 19987071
## 2 Afghanistan 20 00 2666 20595360
## 3 Brazil 19 99 37737 172006362
## 4 Brazil 20 00 80488 174504898
## 5 China 19 99 212258 1272915272
## 6 China 20 00 213766 1280428583El comando unite() literalmente une dos o más columnas. Sus argumentos son: 1) col, el nombre de la nueva columna, 2) la lista de columnas a unir, 3) sep, el caracter que indica cómo concatenar las columnas (pre establecido en "_".)
table_final <- unite(table_final, col=year, century, year, sep="")
table_final## # A tibble: 6 x 4
## country year cases population
## <chr> <chr> <chr> <chr>
## 1 Afghanistan 1999 745 19987071
## 2 Afghanistan 2000 2666 20595360
## 3 Brazil 1999 37737 172006362
## 4 Brazil 2000 80488 174504898
## 5 China 1999 212258 1272915272
## 6 China 2000 213766 1280428583Última actualización: 02-09-2020.
